牛津、EPFL等团队利用Transformer架构开发基于WES数据的体细胞和种系CNV调用程序,准确率为68.7%,召回率为49.6%
 发布于2024-11-05 阅读(0)
发布于2024-11-05 阅读(0)
扫一扫,手机访问

编辑 | 萝卜皮
拷贝数变异(CNV)与多种遗传性疾病的发生有密切关系。为了准确检测CNV,临床上一直在努力研究利用全外显子组测序(WES)数据的方法。尽管最近的算法性能有所提高,但准确检测CNV仍然是一个挑战,因为大多数算法的精度较低,专家策划的黄金标准调用集的召回率甚至更低。
牛津大学(Oxford University)、瑞士洛桑联邦理工学院(EPFL)以及土耳其毕尔肯大学(Bilkent University)近日联合发布了一个名为ECOLE的基于深度学习的WES数据体细胞和种系CNV调用程序。ECOLE的开发旨在提供一种高效准确的方法,用于从WES数据中检测体细胞和种系CNV。这一研究成果有望为基因组学研究和临床应用领域带来重要的突破。
基于Transformer架构的变体,该模型利用高置信度调用对匹配的WGS样本进行学习,以预测每个外显子的CNV。
研究人员利用迁移学习将一小组专家调用,对模型进行进一步训练和微调。ECOLE 首次在人类专家标记数据上取得了优异的表现,准确率达到68.7%,召回率达到49.6%。
与排名第二的最佳方法相比,我们的解决方案在准确率和召回率方面分别提高了18.7%和30.8%。通过使用相同的微调策略,我们的模型ECOLE能够在膀胱癌样本中检测到经过RT-qPCR验证的变异,无需对照样本。
在《Nature Communications》于2024年1月2日发布的一项研究中,名为「ECOLE: Learning to call copy number variants on whole exome sequencing data」的研究成果受到了广泛关注。该研究集中探讨了如何利用全外显子测序数据来准确识别基因拷贝数变异。这项研究的结果对于深入了解基因变异与疾病之间的关联具有重要意义,并为相关领域的研究提供了有价值的参考。

拷贝数变异 (CNV) 是被广泛认识为导致癌症、精神分裂症和自闭症等多种疾病的重要危险因素。在过去十年里,高通量测序 (HTS) 技术一直是检测CNV的标准方法。各种基于全基因组测序 (WGS) 数据的CNV检测算法已经取得了显著的成功,其灵敏度和精确度分别高达96%和97%。
这与处理整个外显子组测序 (WES) 数据的算法形成鲜明对比,后者的精度非常低。WGS 是一个更适合这项任务的平台,因为它不使用引入长度、GC 和参考偏差的靶向探针。另一方面,WES 在临床上更具吸引力,因为它比 WGS 更紧凑、可解释且价格实惠。不幸的是,由于这些限制,WES 技术在 CNV 检测方面的临床应用受到限制。
牛津大学、瑞士洛桑联邦理工学院以及土耳其毕尔肯大学的研究人员,前期开发了一种基于深度学习的抛光方法(deep-learning-based polishing approach),该方法可以使用对匹配的 WGS 样本进行的更值得信赖的调用,来纠正许多最先进的基于 WES 的种系 CNV 调用者的调用。虽然这是向前迈出的重要一步,但在临床使用方面仍然存在瓶颈。
第一个问题是结果的敏感性。抛光器(polisher)只能处理基本算法返回的调用(例如删除)。它要么改变这些调用(例如,重复),要么中和它们(例如,不调用)。虽然这有助于降低错误发现率,但它对灵敏度的影响有限,因为抛光器无法进行新的调用(例如,将无调用转换为删除/重复)。不幸的是,由于性能非常低,灵敏度大多超出了基于 WES 的 CNV 调用域的范围。
第二个问题是,即使是经过优化后的精度性能也受到专家策划的 CNV 调用集的限制,这些调用集被视为黄金地面事实(高达 35%)。这是因为抛光器使用基于 WGS 的自动化 CNV 调用作为模型训练的标签,但这些标签(调用)与人类专家决策相比具有非常不同的分布。
不幸的是,这种手动策划的调用集尺寸非常小,这阻碍了机器学习模型的训练。因此,在人类专家策划的 CNV 调用集上实现高性能的调用者将使基于 WES 的种系 CNV 检测在临床中得到广泛使用。
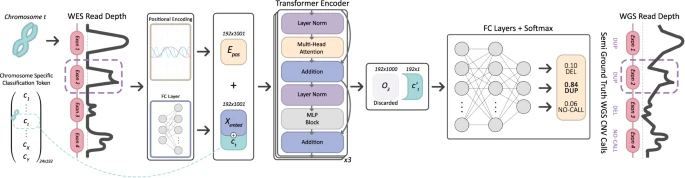
图示:ECOLE 的系统概述。(来源:论文)
因此,该团队又提出了第一个基于深度学习的方法(ECOLE: Exome-based COpy number variation calling LEarner),该方法可以独立学习对 WES 数据执行体细胞和种系 CNV 调用。该模型基于 Transformer 模型的变体,这是自然语言处理领域处理序列数据的最先进方法。
ECOLE 可以处理每个外显子的读取深度信号。它了解需要关注信号的哪些部分以及在哪个上下文(即染色体)中调用 CNV。它使用在匹配的 WGS 样本上获得的高置信度调用(即标签)作为半真实值。
ECOLE 在自动 WGS 调用的基准上显着提高了外显子精确度以及第二最佳方法性能的召回率(分别提高了 13.5% 和 16.6%)。这是唯一具有平衡精度和召回率的方法。
此外,该团队还首次提出使用转移学习并使用少量人类专家标记的样本来微调模型参数。研究人员表明,这种方法在预测人类标签方面的精度和召回率分别提高了约 18% 和约 30%。
同样,他们使用微调方法使 ECOLE 能够使用膀胱癌样本来调用体细胞变异。研究人员证明,他们能够在 16 个膀胱癌样本中的 13 个样本中检测到经过 PCR 验证的拷贝数畸变,而最先进的方法即使在抛光后也只能检测到 2 个样本中经过验证的拷贝数畸变。
该方法具有作为种系和体细胞 CNV 调用者的能力,并通过微调轻松适应疾病和人类专家;研究人员建议 ECOLE 作为扩大外显子组测序技术在 CNV 检测临床应用的可行选择。
ECOLE:https://github.com/ciceklab/ECOLE
论文链接:https://www.nature.com/articles/s41467-023-44116-y
产品推荐
-

售后无忧
立即购买>- DAEMON Tools Lite 10【序列号终身授权 + 中文版 + Win】
-
¥150.00
office旗舰店
-

售后无忧
立即购买>- DAEMON Tools Ultra 5【序列号终身授权 + 中文版 + Win】
-
¥198.00
office旗舰店
-

售后无忧
立即购买>- DAEMON Tools Pro 8【序列号终身授权 + 中文版 + Win】
-
¥189.00
office旗舰店
-

售后无忧
立即购买>- CorelDRAW X8 简体中文【标准版 + Win】
-
¥1788.00
office旗舰店
-
 正版软件
正版软件
- FLOKI币未来价值2024年?FLOKI币在哪个交易所
- FLOKI币价格今日行情$0.00024≈¥0.00174FLOKI币未来价值2024年凭借其坚实的基础和专注的加密社区支持,FLOKI有望在未来获得更大的成功。如果FLOKI项目宣布与其他网络合作,到2024年,FLOKI币的最高价格很可能超过0.00031389美元。如果当前的加密市场继续看涨,我们预计到2024年,FLOKI币的平均价格将保持在约0.000178美元左右。能涨到1美元吗?可能会。许多加密货币分析师和投资者预测FLOKI(FLOKI)将在未来达到0.1美元大关,尽管
- 4分钟前 虚拟货币 区块链 比特币 FLOKI币 0
-
 正版软件
正版软件
- tp钱包领空投地址
- 什么是TP钱包空投地址?TP钱包空投地址是用于接收加密货币空投的专属地址。如何找到您的TP钱包空投地址?打开TP钱包应用程序。选择“资产”选项卡。点击“接收”按钮。在“地址”字段下找到您的空投地址。如何使用您的TP钱包空投地址?拥有空投资格的钱包。将空投地址提供给发放方。等候空投,通常在活动结束或满足条件后分发。
- 14分钟前 0
-
 正版软件
正版软件
- xPet仿盘!Solana链上爆红宠物游戏OKZOO怎么玩?
- 该网站120BtC.coM推出了一款名为「虚拟宠物养成」的游戏OKZOO,这款游戏迅速成为社群话题,总下载量已经冲破21,000次。与xPet类似,OKZOO同样提供浏览器扩展程序存储,并且游戏画面展示在X平台上。OKZOO:Solana上的xPet仿盘OKZOO是由PandaPoo创立的项目,获得了GuildFi、Moby、GameSwift机构的孵化支持,并成功吸引了包括0xSun、JosephYoung、JunPark等天使投资人投资。由于游戏刚于昨日开放注册,许多功能能够预计还在开发中。目前只能先
- 29分钟前 比特币爆仓 区块链游戏 迅雷玩客币 玩客币 区块链怎么赚钱 0
-
 正版软件
正版软件
- 特斯拉Cybertruck自动换挡实测:开启智能驾驶新篇章
- 4月21日消息,近日特斯拉新车型因全面采用屏幕换挡方式而受到部分车主的质疑,认为其操作不太直观与便捷。然而,这背后实际上蕴藏着特斯拉更深远的科技布局。悚车帝最近对特斯拉Cybertruck的自动换挡功能进行了独家实测,揭示了屏幕换挡背后的真正意图。数据车帝(DataCarEmperor)是Cybertruck的车机系统中发现了一个名为“AutoShiftoutofPark”的新功能,这意味着车辆能够通过前后摄像头智能判断应当挂在停车挡还是倒挡。在实际测试中,当Cybertruck处于驻车挡(P挡)且前方有
- 44分钟前 特斯拉 0
-
 正版软件
正版软件
- 比特币为什么可以交易
- 比特币的交易能力源于:区块链技术、去中心化、稀缺性、全球接受度、流动性、匿名性和低交易费用,使其成为一种安全、便捷、有吸引力的交易资产。
- 59分钟前 0
最新发布
-
 1
1
- 阿里追捧的中台,“热度”退了?
- 1856天前
-
 2
2
- Overture设置踏板标记的方法
- 1693天前
-
 3
3
- 思杰马克丁取得CleanMyMac中国区独家发行授权
- 1683天前
-
 4
4
- IBM:20万台Mac让公司职工在工作中更快乐 更多产
- 1881天前
-
 5
5
- 报道称微软一直在悄然游说反对“维修权”立法!
- 1847天前
-
 6
6
- 美国怀疑华为窃取商业机密 华为:身正不怕影子斜
- 1843天前
-
 7
7
- 三星被曝正与联发科接洽 A系列手机有望搭载其5G芯片
- 1858天前
-
 8
8
- 环球墨非完成千万级融资 联合企业集团投资
- 1879天前
-
 9
9
相关推荐
热门关注
-

- Xshell 6 简体中文
- ¥899.00-¥1149.00
-

- DaVinci Resolve Studio 16 简体中文
- ¥2550.00-¥2550.00
-

- Camtasia 2019 简体中文
- ¥689.00-¥689.00
-

- Luminar 3 简体中文
- ¥288.00-¥288.00
-

- Apowersoft 录屏王 简体中文
- ¥129.00-¥339.00